宏基因組binning的原理是什么
這期內(nèi)容當(dāng)中小編將會(huì)給大家?guī)?lái)有關(guān)宏基因組binning的原理是什么,文章內(nèi)容豐富且以專(zhuān)業(yè)的角度為大家分析和敘述,閱讀完這篇文章希望大家可以有所收獲。
創(chuàng)新互聯(lián)是由多位在大型網(wǎng)絡(luò)公司、廣告設(shè)計(jì)公司的優(yōu)秀設(shè)計(jì)人員和策劃人員組成的一個(gè)具有豐富經(jīng)驗(yàn)的團(tuán)隊(duì),其中包括網(wǎng)站策劃、網(wǎng)頁(yè)美工、網(wǎng)站程序員、網(wǎng)頁(yè)設(shè)計(jì)師、平面廣告設(shè)計(jì)師、網(wǎng)絡(luò)營(yíng)銷(xiāo)人員及形象策劃。承接:網(wǎng)站建設(shè)、網(wǎng)站制作、網(wǎng)站改版、網(wǎng)頁(yè)設(shè)計(jì)制作、網(wǎng)站建設(shè)與維護(hù)、網(wǎng)絡(luò)推廣、數(shù)據(jù)庫(kù)開(kāi)發(fā),以高性?xún)r(jià)比制作企業(yè)網(wǎng)站、行業(yè)門(mén)戶(hù)平臺(tái)等全方位的服務(wù)。
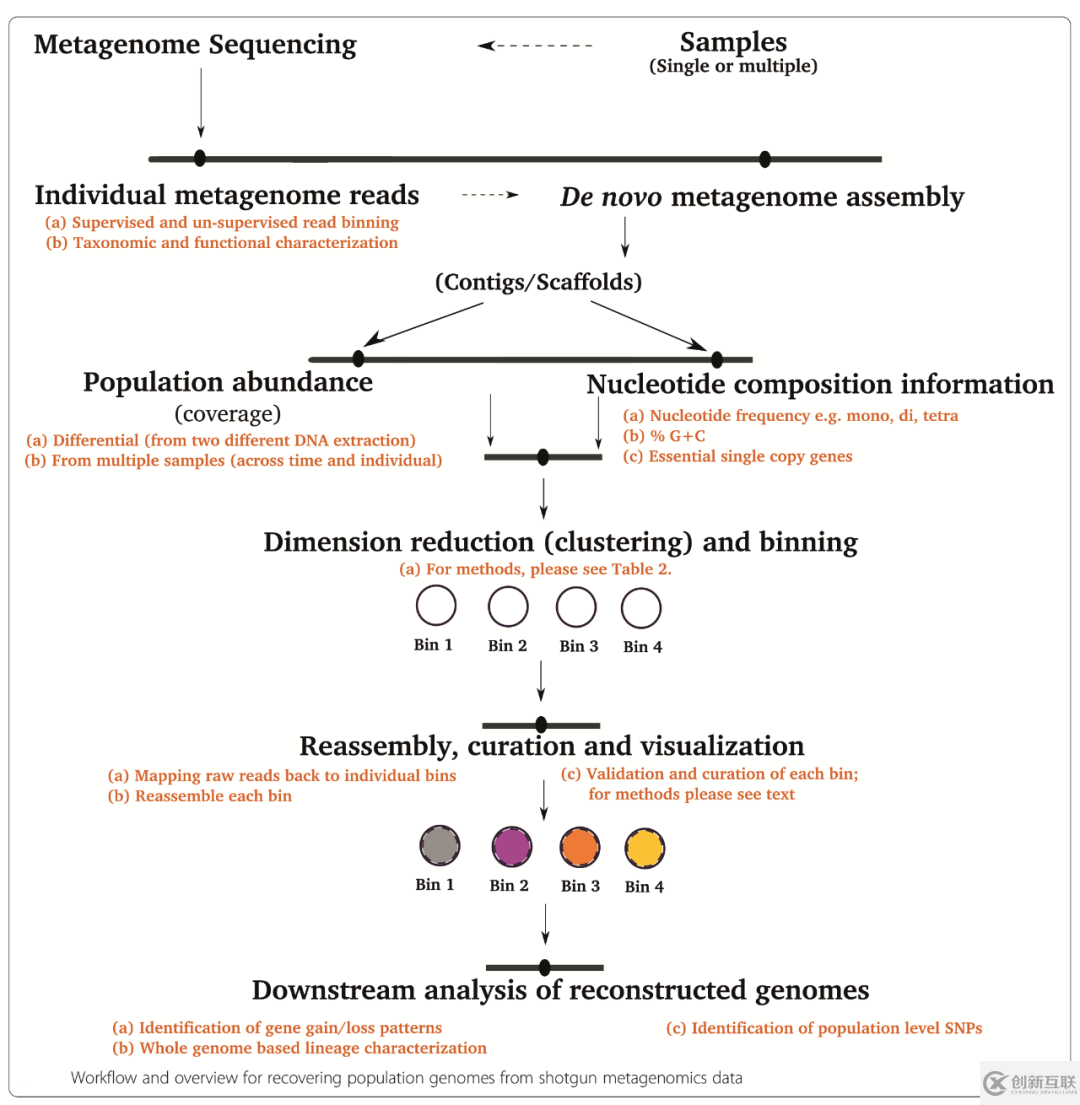
在宏基因組中分離單基因組,可利用序列特征或序列組裝信息,常見(jiàn)的可用信息主要有以下幾種:
a.根據(jù)核酸使用頻率(通常是四核苷酸頻率)、GC含量和必需的單拷貝基因等基因組特征;
b.根據(jù)contig序列的覆蓋度coverage信息;
c.根據(jù)測(cè)序數(shù)據(jù)的kmer豐度信息;
d.根據(jù)序列在不同樣品的共出現(xiàn)規(guī)律(co-abundance patternsacross multiple samples);
e.將序列map到數(shù)據(jù)庫(kù)的參考序列所獲得的注釋信息,也即物種binning。
根據(jù)所使用的序列數(shù)據(jù)不同,binning策略可分為三種:基于組裝前的clean reads,基于組裝后的contigs,基于注釋的基因genes。
⑴基于reads binning
環(huán)境樣本中微生物的豐度不同,其基因組kmer的期望深度也不同,根據(jù)kmer豐度可以直接對(duì)reads進(jìn)行聚類(lèi),將屬于不同基因組的reads分離開(kāi)來(lái)。其優(yōu)勢(shì)是可以聚類(lèi)出宏基因組中豐度非常低的物種,而且可以分離系統(tǒng)發(fā)育關(guān)系很近的物種。考慮到在宏基因組組裝中reads利用率很低,單樣品5Gb測(cè)序量情況下,環(huán)境樣品組裝reads利用率一般只有10%左右,腸道樣品或極端環(huán)境樣品組裝reads利用率一般能達(dá)到30%,這樣很多物種,尤其是低豐度的物種的reads沒(méi)有被沒(méi)有被組裝出來(lái),沒(méi)有體現(xiàn)在contig中而被浪費(fèi),因此基于reads binning才有可能得到低豐度的物種基因組的的測(cè)序數(shù)據(jù),在實(shí)際研究中基于reads binning的LSA(Latent Strain Analysis)方法可以聚類(lèi)出豐度低到0.00001%的物種,并且對(duì)同一物種中的不同菌株的敏感性很強(qiáng)[2]。
⑵基于genes binning
在宏基因組做完序列組裝和基因預(yù)測(cè)之后,把所有樣品中預(yù)測(cè)到的基因混合在一起,去冗余得到unique genes集合,根據(jù)gene在各個(gè)樣品中的豐度變化模式,計(jì)算gene之間的相關(guān)性,利用這種相關(guān)性進(jìn)行聚類(lèi)。利用這種策略進(jìn)行binning得到的bins可稱(chēng)為CAG(co-abundance genegroups),包含有700個(gè)以上的gene的CAG稱(chēng)為MGS(metagenomic species),CAG可用進(jìn)行關(guān)聯(lián)分析,MGS可用進(jìn)行后續(xù)的單菌組裝[3]。當(dāng)然根據(jù)具體的聚類(lèi)算法和相關(guān)性系數(shù)的不同,對(duì)genes binning得到的bins的叫法也不同,除以上外還有MLG(metagenomic linkage groups)、MGC(metagenomic clusters)和MetaOTUs(metagenomic operational taxonomicunits)等,同時(shí),MLG, MGC, MGS和MetaOTUs物種注釋的標(biāo)準(zhǔn)也是不一樣的。
目前已發(fā)表的宏基因組關(guān)聯(lián)分析(MWAS)和多組學(xué)聯(lián)合分析文章中,宏基因組binning很多都用genes binning方法,尤其是疾病的MWAS研究中基本都用genes binning[4]。這種方法的優(yōu)勢(shì)是基于genes豐度變化模式進(jìn)行binning可操作性比較強(qiáng),過(guò)程比較簡(jiǎn)單,可復(fù)制性強(qiáng),對(duì)計(jì)算機(jī)資源消耗比較低。
⑶基于contigs binning
在宏基因組做完序列組裝之后,將所有reads序列map到contigs上獲得contig覆蓋率,再綜合GC含量、核算組成等信息對(duì)contig進(jìn)行聚類(lèi),將屬于不同基因組的contig序列分開(kāi)。contig binning目前應(yīng)用十分廣泛,最常用的就是用于組裝單物種基因組,目前已經(jīng)有多種基于contig binning的軟件[1],對(duì)于豐度較高的物種contigs binning效果較好,但是目前也有些缺陷或者說(shuō)還有很多可提升的空間,例如對(duì)核酸組成信息的利用,開(kāi)發(fā)得就不夠充分,四堿基使用頻率因簡(jiǎn)單而被廣泛使用和接受,但現(xiàn)在已有研究表明k-mer豐度信息也是很好的種系特征,同時(shí)越長(zhǎng)的k-mer含有越多的信息,還有基因和參考基因組間的同源關(guān)系也是有價(jià)值的種系信號(hào),但這些都還沒(méi)有被自動(dòng)化的binning軟件整合。
上述就是小編為大家分享的宏基因組binning的原理是什么了,如果剛好有類(lèi)似的疑惑,不妨參照上述分析進(jìn)行理解。如果想知道更多相關(guān)知識(shí),歡迎關(guān)注創(chuàng)新互聯(lián)行業(yè)資訊頻道。
分享題目:宏基因組binning的原理是什么
網(wǎng)站鏈接:http://m.newbst.com/article40/jocdeo.html
成都網(wǎng)站建設(shè)公司_創(chuàng)新互聯(lián),為您提供網(wǎng)站收錄、做網(wǎng)站、面包屑導(dǎo)航、商城網(wǎng)站、手機(jī)網(wǎng)站建設(shè)、
聲明:本網(wǎng)站發(fā)布的內(nèi)容(圖片、視頻和文字)以用戶(hù)投稿、用戶(hù)轉(zhuǎn)載內(nèi)容為主,如果涉及侵權(quán)請(qǐng)盡快告知,我們將會(huì)在第一時(shí)間刪除。文章觀點(diǎn)不代表本網(wǎng)站立場(chǎng),如需處理請(qǐng)聯(lián)系客服。電話(huà):028-86922220;郵箱:631063699@qq.com。內(nèi)容未經(jīng)允許不得轉(zhuǎn)載,或轉(zhuǎn)載時(shí)需注明來(lái)源: 創(chuàng)新互聯(lián)

- 外貿(mào)建站域名該怎么選? 2015-06-08
- 為什么要選擇外貿(mào)建站? 2015-04-27
- 外貿(mào)建站seo推廣除了內(nèi)容,頁(yè)面鏈接也很重要 2016-08-19
- 外貿(mào)建站中的那些細(xì)節(jié)影響著網(wǎng)站流量? 2015-04-24
- 外貿(mào)建站選擇香港主機(jī)都有哪些優(yōu)勢(shì)? 2022-10-10
- 外貿(mào)建站沒(méi)有你想的那么簡(jiǎn)單 2015-04-16
- 做外貿(mào)建站為什么要選穩(wěn)定快速安全的海外主機(jī)? 2015-04-29
- 外貿(mào)建站和普通網(wǎng)站建設(shè)的區(qū)別 2022-10-22
- 詮釋網(wǎng)站排名的高低與流量多少之間的關(guān)系 2022-06-24
- 按外貿(mào)建站域名五原則挑選老外喜歡的域名 2015-05-01
- 外貿(mào)建站的推廣方式有哪些 2016-04-18
- 越秀區(qū)小北路外貿(mào)建站,越秀區(qū)小北路外貿(mào)網(wǎng)站建設(shè)公司 2016-04-06